Offerta tesi
09-17-19- Tesi di Laurea Magistrale in Genetica e Biologia Molecolare, Biologia e Tecnologie Cellulari, Neurobiologia.
- Presso RNALAB@IBPM (Sapienza Università di Roma, Edificio CU026) sono disponibili due posti per lo svolgimento di tesi magistrali sperimentali.
- Le attività si concentreranno sulla caratterizzazione funzionale di lunghi RNA non codificanti in modelli fisiopatologici in vitro del sistema nervoso, nell’ambito delle seguenti linee di ricerca:
1) Ruolo funzionale di long non-coding RNA in tumori cerebellari pediatrici (parte del progetto PNRR: MEDFER).
Riferimenti: doi: 10.3389/fcell.2020.00275; doi: 10.3390/cancers13153853.
2) Ruolo dei long non-coding RNA nell’attività e nella degenerazione di motoneuroni (parte del progetto PRIN 2022: NOMeN).
Riferimenti: doi: 10.1038/s41419-020-02738-w; doi: 10.1038/s41419-023-06196-y.
- Obiettivi: gli studenti avranno l'opportunità di acquisire competenze in tecniche di biologia molecolare e biochimica dell’RNA (clonaggio, genetica inversa, analisi di espressione genica, analisi -omiche, interazioni, imaging) e di biologia cellulare (colture cellulari, differenziamento, fenotipizzazione).
- Eleggibilità: priorità sarà accordata a candidati iscritti al primo anno di Laurea Magistrale.
- Inizio: preferibilmente entro novembre-dicembre 2024.
- Selezione: inviare CV dettagliato del percorso accademico e completo di votazioni, a Pietro Laneve. È prevista un’eventuale fase di colloquio orale.
- Tesi di Laurea Magistrale in Discipline di area biologica, genetica, antropologia, archeologia
Presso il laboratorio di Genetica delle Popolazioni, Edificio di Genetica CU022, sono disponibili due progetti di tesi sperimentale sui seguenti argomenti:
Progetto1: Analisi del DNA antico (aDNA) di individui dell’Età del Rame dell’Italia centro-meridionale
Progetto2: Analisi archeogenetica di siti tardo-antichi al confine tra i territori longobardi e bizantini nell'Italia centrale
Relatori: Eugenia D’Atanasio, IBPM-CNR; Beniamino Trombetta, Dipartimento di Biologia e Biotecnologie «Charles Darwin», Sapienza
Candidature: Inviare agli indirizzi indicati sopra un CV completo di: votazione di laurea triennale, votazioni e relativi CFU degli esami di Laurea Triennale, votazioni e relativi CFU agli esami di Laurea Magistrale (se già sostenuti).
- Tesi di Laurea Magistrale in Genetica e Biologia Molecolare, in Biologia e Tecnologie Cellulari, Magistrale in Biotecnologie e Genomica per l'industria e l'ambiente
Nel laboratorio di Biologia Molecolare delle Piante (PajoroLab) sono disponibili due posti per lo svolgimento di tesi magistrali sperimentali.
L’attività di ricerca è incentrata sull’approccio di biologia traslazionale per l’aumento della produttività della pianta.
I progetti di tesi si inseriscono nei seguenti progetti:
1) StemFlow: Funzione dei geni che regolano la transizione fiorale nel determinare l’architettura della pianta (Arabidopsis).
2) Rucol-ITA. Miglioramento genetico di Diplotaxis tenuifolia per ritardare la fioritura.
Lo studente avrà l'opportunità di apprendere le tecniche di biologia molecolare, fisiologia vegetale, ingegneria genetica (TEA), potrà inoltre acquisire familiarità con dati NGS e data visualization.
Nel laboratorio sono attivati annualmente tirocini extra-curricolari per laureati Sapienza.
Per informazioni rivolgersi a: Alice Pajoro
- Tesi di Laurea Magistrale in Genetica e Biologia Molecolare, in Biologia e Tecnologie Cellulari, in Biotecnologie e Genomica per l'industria e l'ambiente
Presso il laboratorio di Acidi Nucleici sono disponibili tre posti per lo svolgimento di tesi sperimentali magistrali. L’attività del nostro laboratorio è incentrata sui seguenti argomenti:
1) Network regolativi che controllano la crescita e lo sviluppo della pianta in risposta a segnali di stress ambientali abiotici (temperatura, salinità, colture intensive).
2) Ruolo dei fattori di trascrizione HD-Zip II nello sviluppo vegetale (meristema apicale e radicale, fioritura, germinazione del seme).
Lo studente avrà l'opportunità di apprendere le tecniche di biologia molecolare, di biologia celllulare e di fisiologia applicate allo studio di specie vegetali. Le specie utilizzate sono Arabidopsis thaliana e brassicacee, Lycopersicum esculentum, Lactuca sativa.
Per informazioni rivolgersi a: Monica Carabelli, Giovanna Sessa e Massimiliano Sassi
- Tesi di Laurea Magistrale in Genetica e Biologia Molecolare
Presso il laboratorio di Genetica Umana è disponibile un posto per lo svolgimento di tesi sperimentale sul seguente argomento:
"Studio della lunghezza dei telomeri dei Leucociti e degli SnoRNA come biomarcatori di malattie neurodegenerative."
Lo studente avrà l'opportunità di apprendere le tecniche di base di biologia molecolare.
Per informazioni rivolgersi a: Daniela Scarabino
- Tesi di laurea triennale in Scienze Biologiche e Biotecnologie:
- Tesi di laurea magistrale in Genetica e Biologia Molecolare o Biologia e Tecnologie Cellulari:
Presso il laboratorio di Genetica, Dip di Biologia e Biotecnologie Darwin, sede di via degli Apuli, sono disponibilii:
- un posto per lo svolgimento di una tesi sperimentale di laurea triennale
- un posto per lo svolgimento di una tesi sperimentale magistrale
L'argomento riguarderà lo "Studio dei telomeri umani come bersaglio nella terapia anti-tumorale". Il candidato avrà l'opportunità di apprendere le tecniche di base di biologia cellulare e molecolare.
Per informazioni rivolgersi a: Erica Salvati
- Tesi di laurea triennale in Scienze Biologiche e Biotecnologie.
Presso il laboratorio di Genetica, Dip di Biologia e Biotecnologie Darwin, sede di via degli Apuli, è disponibile un posto per lo svolgimento di una tesi sperimentale.
Argomento: Colture cellulari umane e saggi di geni e molecole di interesse terapeutico
Per informazioni: Patrizia Lavia
- Tesi di Laurea Magistrale in: Chimica e Tecnologia Farmaceutiche, in Farmacia, in Biotecnologie Farmaceutiche, in Biologia e Tecnologie Cellulari, in Genetica e Biologia Molecolare
Presso il laboratorio di Biologia Molecolare, Dip di Scienze Biochimiche, è disponibile un posto per lo svolgimento di tesi sperimentale sul seguente argomento:
Argomento tesi: "Biochimica del metabolismo dell’acido solfidrico e sue implicazioni fisiopatologiche"
Per informazioni rivolgersi a: Alessandro Giuffrè
- Tesi di Laurea Magistrale in Genetica e Biologia Molecolare
Presso il laboratorio di Acidi Nucleici, Dip di Biologia e Biotecnologie Darwin, sede di via degli Apuli, è disponibile un posto per lo svolgimento di una tesi sperimentale
Argomento: “Studio del ruolo di proteine di legame all'RNA nel metabolismo e l'attivita' dei microRNA”
Il candidato avrà l'opportunità di apprendere tecniche di biologia molecolare, biologia cellulare, analisi bioinformatica
Per informazioni: Cecilia Mannironi
Davide Valente CV-IT
03-27-19Davide Marzi CV-IT
02-21-19Paola Rovella CV-IT
02-18-192015: Laurea Magistrale in Biologia, Indirizzo bio-molecolare, Università della Calabria.
2010: Laurea Triennale in Scienze Biologiche, Università della Calabria.
Esperienze post-Lauream
Gennaio 2020- in corso: Assegnista di ricerca, Istituto di Biologia e Patologia Molecolari (IBPM), CNR
Progetto PRIN-MIUR: Microtubule and centrosome dynamics, from Omics to neurodevelopmental disorders of Central Nervous System
Marzo 2018- Settembre 2019: Borsista, Istituto di Biologia e Patologia Molecolari (IBPM), CNR
Progetto AIRC: The multifaceted Aurora-A/TPX2 complex in chromosome stability, tumorigenesis and anti-cancer therapy
Giugno 2017-Novembre 2017: Borsista, Istituto Pasteur Italia-Fondazione Cenci Bolognetti, c/o Dipartimento di Biologia e Biotecnologie “Charles Darwin, Università La Sapienza, Roma.
Progetto: Analisi della citochinesi in seguito a knock-down di proteine cromatiniche in cellule umane.
Ottobre 2016-Maggio 2017: Collaborazione con Bionova Technologies s.r.l. c/o Centro di riferimento di microscopia Nikon per l’Italia Centro-Sud, IBPM-CNR, e Dipartimento di Biologia e Biotecnologie, Università La Sapienza, Roma.
Tematica: Valutazione e sviluppo di nuove applicazioni di software di acquisizione ed analisi di immagini in microscopia.
Marzo 2016-Settembre 2016: Tirocinante post-lauream, IBPM-CNR
Progetto AIRC: Farmacogenomica: metodiche per lo studio di farmaci anti-mitotici e modulazione da parte di geni endogeni in cellule tumorali.
Pubblicazioni
- Bellisai C.+, Sciamanna I.+, Rovella P., Giovannini D., Baranzini M., Pugliese G.M., Zeya Ansari M.S., Milite C., Sinibaldi-Vallebona P., Cirilli R., Sbardella G., Pichierri P., Trisciuoglio D., Lavia P., Serafino A., Spadafora C. Reverse transcriptase inhibitors promote the remodeling of nuclear architecture and induce autophagy in prostate cancer cells.
Cancer Letters. (2020) S0304-3835(20)30098-7
-Verrico A., Rovella P., Di Francesco L., Damizia M., Sasah Staid D., Le Pera L., Schininà M.E., and Lavia P. Importin-ß/karyopherin-ß1 modulates mitotic microtubule function and taxane sensitivity in cancer cells via its nucleoporin-binding region.
Oncogene. (2019) 10.1038/s41388-019-0989-x
-La Regina G., Bai R., Coluccia A., Naccarato V., Famiglini V., Verrico A., Rovella P., Mazzoccoli C., Da Pozzo E., Cavallini C., Martini C., Vultaggio S., Dondio G., Varasi M., Mercurio C., Hamel E., Lavia P and Silvestri R. New 6- and 7-heterocyclyl-1H-indole derivatives as potent tubulin assembly and cancer cell growth inhibitors.
Eur J Med Chem. (2018) 152:283-297
-Di Francesco L.+,Verrico A.+, Asteriti IA, Rovella P., Cirigliano P., Guarguaglini G., Schininà M.E. and Lavia P. Visualization of human karyopherin beta-1/importin beta-1 interactions with protein partners in mitotic cells by co-immunoprecipitation and proximity ligation assays.
Sci Rep. (2018) 8:1850.
-Di Cesare E., Verrico A., Miele A., Giubettini M., Rovella P., Coluccia A., Famiglini V., La Regina G., Cundari E., Silvestri R. and Lavia P. Mitotic cell death induction by targeting the mitotic spindle with tubulin-inhibitory indole derivative molecules.
Oncotarget. (2017) 8:19738-19759.
Keywords: Recettori del trasporto nucleare, mitosi, farmaci anti-mitotici, microscopia
Francesca Sardina CV-IT
02-15-192015: Dottorato in Biotecnologie Mediche, Università degli Studi di Siena.
2009: Laurea Specialistica in Biotecnologie per l’ Industria e la Ricerca Scientifica, Università degli Studi di Palermo.
2007: Laurea in Biotecnologie, Università degli Studi di Palermo.
POSIZIONE E ATTIVITA’ DI RICERCA
2022-ad oggi:Borsista di Ricerca, CNR-Istituto di Biologia e Patologia Molecolari (IBPM), Roma.
Progetto di ricerca (AFM-Telethon) “Phenotypic evaluation of microtubules network as prognostic and predictive marker in response to spastin elevating approaches in Hereditary Spastic Paraplegia type 4”.
2019-2022:Assegnista di Ricerca, CNR-Istituto di Biologia e Patologia Molecolari (IBPM), Roma. Progetto di ricerca (AFM-Telethon): “The HIPK2 kinase/spastin axis in Hereditary Spastic Paraplegia.
2018-2019:Borsista di ricerca post-dottorale (EMBO Short-term fellow), Sheffield Institute for Translational Neuroscience, University of Sheffield (UK).
Progetto di ricerca: “The HIPK2 kinase/spastin axis in Hereditary Spastic Paraplegia (HSP)”.
2017-2018:Assegnista di Ricerca, CNR-Istituto di Biologia e Patologia Molecolari (IBPM), Roma.
Progetto di ricerca (Telethon): “The role of HIPK2 kinase in regulation of the spastin protein in proliferating cells and in neurons”.
2014-2016:Borsista di ricerca post-dottorale, Dipartimento di Medicina Molecolare dell’ Università “La Sapienza”, Roma.
Progetto di ricerca (AIRC): “Role of the DNA damage response in hedgehog-MYCN dependent proliferation of cerebellar stem cells and granule precursor cells”.
2010-2014:Dottoranda, Dipartimento di Medicina Molecolare dell’ Università di Roma “La Sapienza”, Roma.
Progetto di dottorato: “The MRN complex controls MYCN-dependent replication stress and is a potential therapeutic target for MYCN-amplified neuroblastoma”.
2009-2010:Tirocinante post-laurea, CNR-Istituto di Biomedicina e Immunologia Molecolare (IBIM), Palermo.
Progetto di ricerca: “The effects of the novel NF-?B inhibitor dehydroxymethylepoxyquinomicin (DHMEQ) in combination with the selective COX-2 inhibitor celecoxib, in human hepatoma cells”.
PREMI DI RICERCA
2021:A.I.Vi.P.S ETS premio di ricerca “Microtubule cytoskeleton organization as biomarker to develop new therapeutic perspectives for Hereditary Spastic Paraplegia type 4 (HSP-SPG4)”
2021:AFM-Telethon Postdoctoral fellowship “Phenotypic evaluation of microtubules network as prognostic and predictive marker in response to spastin elevating approaches in Hereditary Spastic Paraplegia type 4” (rif N°23786).
2021:Adolf-Struempell-Prize per il manoscritto dal titolo Sardina et al., LSA 2020; doi: 10.26508/lsa.202000799.
PUBBLICAZIONI
1-Sardina F (corresponding author), Valente D, Fattorini G, Cioffi E, Zanna GD, Tessa A, Trisciuoglio D, Soddu S, Santorelli FM, Casali C, Rinaldo C. New cellular imaging-based method to distinguish the SPG4 subtype of hereditary spastic paraplegia. Eur J Neurol. 2023 Jun;30(6):1734-1744. doi: 10.1111/ene.15756.
2-Sardina F, Conte A, Paladino S, Pierantoni GM, Rinaldo C. HIPK2 in the physiology of nervous system and its implications in neurological disorders. Biochim Biophys Acta Mol Cell Res. 2023 Jun;1870(5):119465. doi: 10.1016/j.bbamcr.2023.119465. Epub 2023 Mar 20. PMID: 36935052; PMCID: PMC10236203.
3-Sardina F (co-corresponding author), Pisciottani A, Ferrara M, Valente D, Casella M, Crescenzi M, Peschiaroli A, Casali C, Soddu S, Grierson AJ, Rinaldo C. Spastin recovery in hereditary spastic paraplegia by preventing neddylation-dependent degradation. Life Sci Alliance. 2020 Oct 26;3(12):e202000799. doi: 10.26508/lsa.202000799.
4-Sardina F, Monteonofrio L, Ferrara M, Magi F, Soddu S, Rinaldo C. HIPK2 Is Required for Midbody Remnant Removal Through Autophagy-Mediated Degradation.Front Cell Dev Biol. 2020 Sep 15;8:572094. doi: 10.3389/fcell.2020.572094.
5-Petroni M, Sahùn Roncero M, Ramponi V, Fabretti F, Nicolis Di Robilant V, Moretti M, Alfano V, Corsi A, De Panfilis S, Giubettini M, Di Giulio S, Capalbo C, Belardinilli F, Coppa A, Sardina F, Colicchia V, Pedretti F, Infante P, Cardinali B, Tessitore A, Canettieri G, De Smaele E, Giannini G. SMO-M2 mutation does not support cell-autonomous Hedgehog activity in cerebellar granule cell precursors. Sci Rep. 2019 Dec 23;9(1):19623. doi: 10.1038/s41598-019-56057-y.
6-Pisciottani A, Biancolillo L, Ferrara M, Valente D, Sardina F, Monteonofrio L, Camerini S, Crescenzi M, Soddu S, Rinaldo C. HIPK2 Phosphorylates the Microtubule-Severing Enzyme Spastin at S268 for Abscission. Cells. 2019 Jul 5;8(7):684. doi: 10.3390/cells8070684.
7-Petroni M, Sardina F (co-first author), Infante P, Bartolazzi A, Locatelli E, Fabretti F, Di Giulio S, Capalbo C, Cardinali B, Coppa A, Tessitore A, Colicchia V, Sahùn Roncero M, Belardinilli F, Di Marcotullio L, Soddu S, Comes Franchini M, Petricci E, Gulino A, Giannini G. MRE11 inhibition highlights a replication stress-dependent vulnerability of MYCN-driven tumors.Cell Death Dis. 2018 Aug 30;9(9):895. doi: 10.1038/s41419-018-0924-z.
8-Colicchia V, Petroni M, Guarguaglini G, Sardina F, Sahún-Roncero M, Carbonari M, Ricci B, Heil C, Capalbo C, Belardinilli F, Coppa A, Peruzzi G, Screpanti I, Lavia P, Gulino A, Giannini G. PARP inhibitors enhance replication stress and cause mitotic catastrophe in MYCN-dependent neuroblastoma.Oncogene. 2017 Aug 17;36(33):4682-4691. doi: 10.1038/onc.2017.40.
9-Petroni M, Sardina F, Heil C, Sahún-Roncero M, Colicchia V, Veschi V, Albini S, Fruci D, Ricci B, Soriani A, Di Marcotullio L, Screpanti I, Gulino A, Giannini G. The MRN complex is transcriptionally regulated by MYCN during neural cell proliferation to control replication stress.Cell Death Differ. 2016 Feb;23(2):197- 206. doi: 10.1038/cdd.2015.81.
10-Cervello M, Bachvarov D, Cusimano A, Sardina F, Azzolina A, Lampiasi N, Giannitrapani L, McCubrey JA, Montalto G. COX-2-dependent and COX-2-independent mode of action of celecoxib in human liver cancer cells. OMICS. 2011 Jun;15(6):383-92. doi: 10.1089/omi.2010.0092.
Premi
09-20-18GIUGNO 2024
Premio della RNA society per il miglior poster nelle ricerche sull’RNA nel Simposio SIBBM 2024 ”The time of Molecular Biology: development, homeostasis and aging”
Trento - 17-19 giugno 2024 https://sibbm2024.azuleon.org/
a Silvia Gasparini
Documents: HL-POSTER SIBBM202
GENNAIO 2024
Premio 2023 a giovani ricercatrici/ ricercatori per la rilevanza dei risultati conseguiti
Dipartimento di Scienze Biomediche del CNR
| Eugenia D'atanasio |
 |
Anna Frappaolo |
|
Premio per la tematica: Scienze EUGENIA D’ATANASIO Analisi genetica del DNA fetale circolante 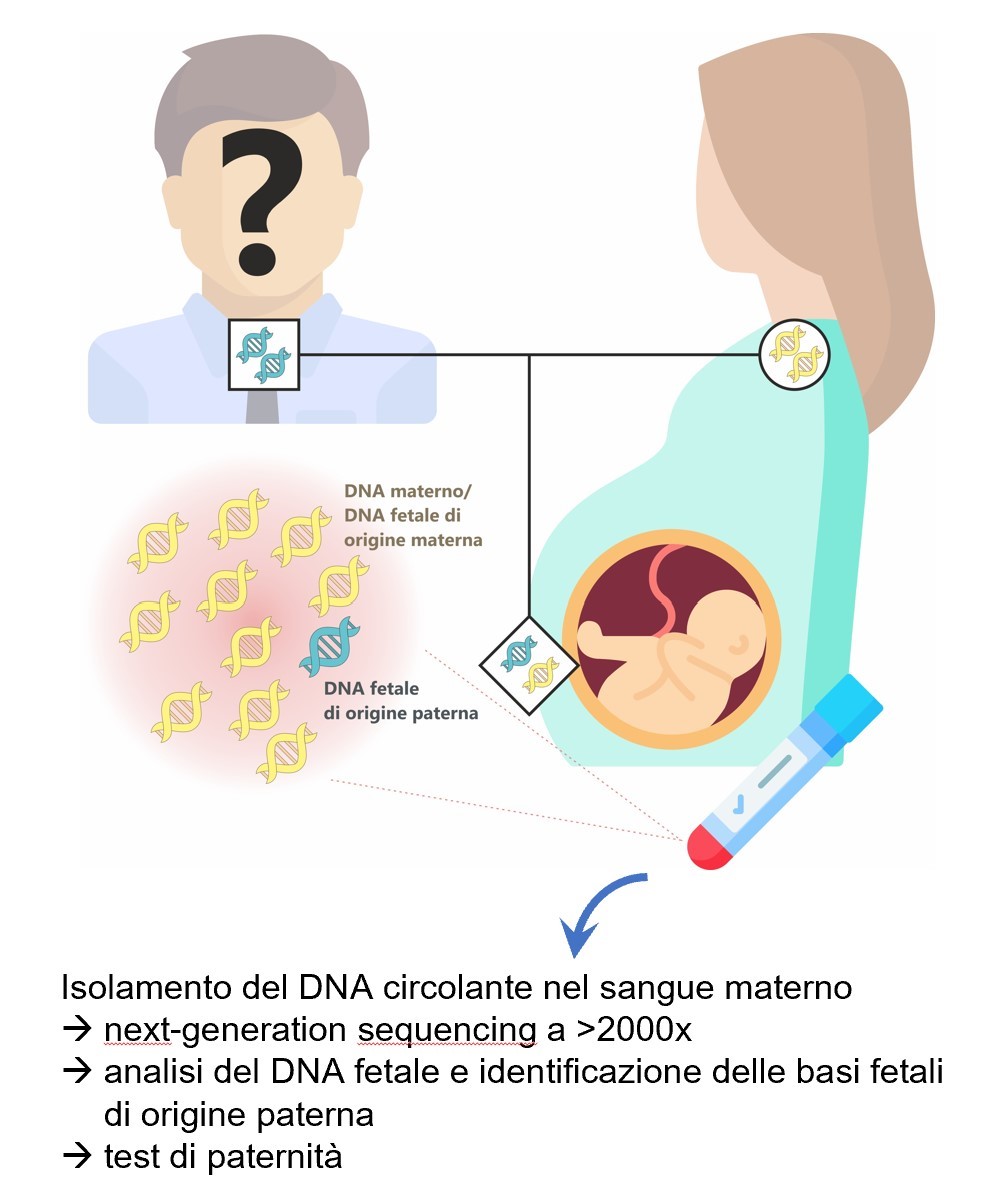 |
Premio per la tematica: Aspetti molecolari, cellulari ANNA FRAPPAOLO Analisi dei meccanismi molecolari che coinvolgono 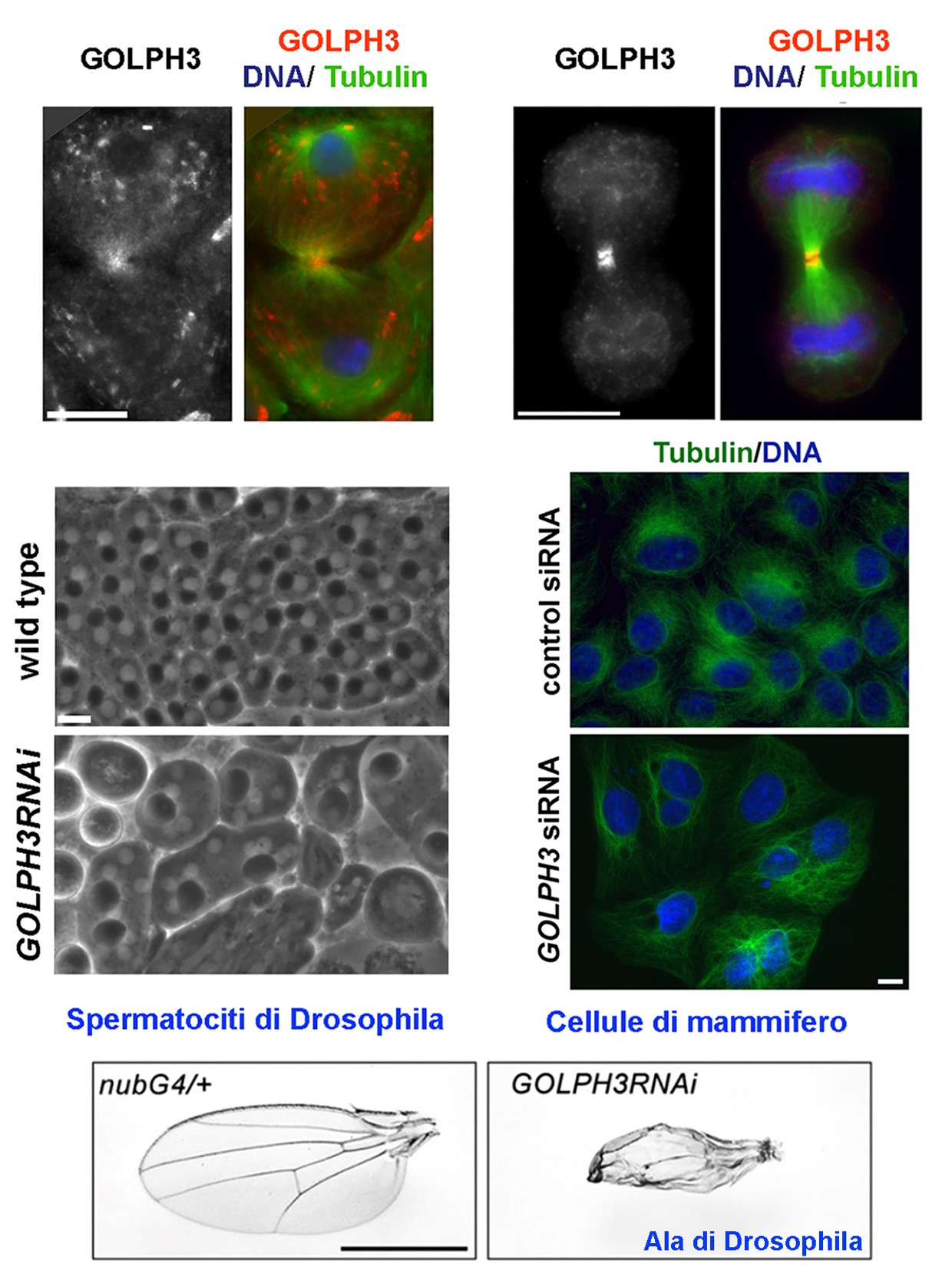 |
DICEMBRE 2023
Borsa della Fondazione Veronesi
https://www.fondazioneveronesi.it/la-fondazione/news-dalla-fondazione/i-vincitori-dei-bandi-2024-di-fondazione-veronesi
a Francesca Sardina
Programma di ricerca: "Targeting spastin by drug repurposing as novel therapeutic strategy to treat glioblastoma"
MARZO 2023
Il Premio “RomaRose - Non solo 8 marzo”, organizzato dalla Presidenza dell’Assemblea capitolina, è nato per premiare l’impegno, il merito e il talento delle donne. A Rossella Lucà è andato il riconoscimento per i suoi studi sulla progressione metastatica dei tumori sviluppando modelli preclinici altamente innovativi in campo oncologico.
Per informazioni:
rossella.luca@cnr.it
FEBBRAIO 2023
Borsa della Fondazione Veronesi
https://www.fondazioneveronesi.it/ricerca/bandi
a Danila Del Rio
Programma di ricerca: "Dissecting how tumor-derived endothelin-1 recruits and activates fibroblasts to support matrix remodelling and tumor aggressiveness in high-grade serous ovarian cancer (HG-SOC)"

OTTOBRE 2021

2021 Adolf Struempell Prize
A Francesca Sardina
Per il manoscritto "Spastin recovery in hereditary spastic paraplegia by preventing neddylation-dependent degradation," Life Sci Alliance. 2020 Oct 26; 3 (12): e202000799 doi: 10.26508 / lsa.202000799”
https://www.hsp-info.de/en/news/news.html?tx_news_pi1%5Bnews%5D=605&tx_news_pi1%5Bcontroller%5D=News&tx_news_pi1%5Baction%5D=detail&cHash=10789b3cdd942adb327338b68800a96e
SETTEMBRE 2021


2021 Research Award da AiViPS ETS e ASL HSP France
A Francesca Sardina
Per il Progetto “Microtubule cytoskeleton organization as biomarker to develop new therapeutic perspectives for Hereditary Spastic Paraplegia type 4 (HSP-SPG4)”
https://www.aivips.it/news/news/197-progetto-di-ricerca-condiviso-italia-francia-su-spg4
A Venturina Stagni
Per il Progetto “Nuovi bersagli molecolari per la fibrodisplasia ossificante progressiva: la via autofagica è coinvolta? ”
https://aws.telethon.it/cosa-facciamo/ricerca/ricercatori/
https://aws.telethon.it/cosa-facciamo/ricerca/progetti-finanziati/nuovi-bersagli-molecolari-per-la-fibrodisplasia-ossificante-progressiva-la-via-autofagica-coinvolta
SETTEMBRE 2021

2021 Post-doctoral fellowship da AFM-Telethon
A Francesca Sardina
Per il Progetto “Phenotipic evaluation of microtubules network as prognostic and predictive marker in response to spastin elevating approaches in Hereditary Spastic Paraplegia type 4”
http://www.afm-telethon.com/research/calls-for-proposals/current-calls-for-proposals.html
SETTEMBRE 2021

2021 Post-doctoral fellowship da AFM-Telethon
A Valeria Bianconi
Per il Progetto “Deciphering the role of Prdm16-mediated H3K9 methylation in the control of Fibro-Adipogenic Progenitors identity and skeletal muscle repair”
http://www.afm-telethon.com/research/calls-for-proposals/current-calls-for-proposals.html
SETTEMBRE 2021
 AFM-TELETHON
AFM-TELETHONA Elisabetta Falvo
Per il Progetto "Personalized non-invasive nanotherapy of Crouzon syndrome through FGFR2 gene knock-down by recombinant human ferritin-based targeted siRNA delivery"
http://www.afm-telethon.com/research/calls-for-proposals/current-calls-for-proposals.html
FISR 2020 COVID
A Corinna Giorgi (PI; IBPM_CNR); Pietro Laneve (IBPM_CNR); Elisa Caffarelli (IBPM_CNR); Silvia Galardi (Univ. di Roma Tor Vergata)
Per il Progetto "ViRAL: una nuova strategia di screening per la rapida identificazione di farmaci contro virus a RNA"
https://www.mur.gov.it/it/atti-e-normativa/decreto-direttoriale-n-1049-del-30-4-2021
MAGGIO 2020: PRESTIGIOSI RICONOSCIMENTI ALLE RICERCHE IBPM

Race to Cure Sarcoma Research Award
a Chiara Mozzetta
Per il progetto “Unwinding new therapeutic opportunities in rhabdomyosarcoma: the role of RNA helicase DDX5”
https://www.curesarcoma.org/sfa-announces-2020-research-grant-awards/

Borsa di Ricerca 2020 della Associazione Nazionale Atassia Telangectasia
a Venturina Stagni
Per il progetto “Targeting autophagy as a potential therapeutic approach for Ataxia Telangiectasia (AT)”
https://www.associazione-at.it/vincitori-borse-di-studio-anat-2020/
Luglio 2019: Due giovani IBPM vincono due finanziamenti ”Torno subito” della Regione Lazio

|
 |
Linea GREEN ECONOMY
- “Una tecnologia innovativa che ottimizza l'uso delle felci per la detossificazione di suoli contaminati da arsenico” MAURA CARDARELLI
- “High content imaging per la validazione di farmaci anti-mitotici” FRANCESCA DEGRASSI
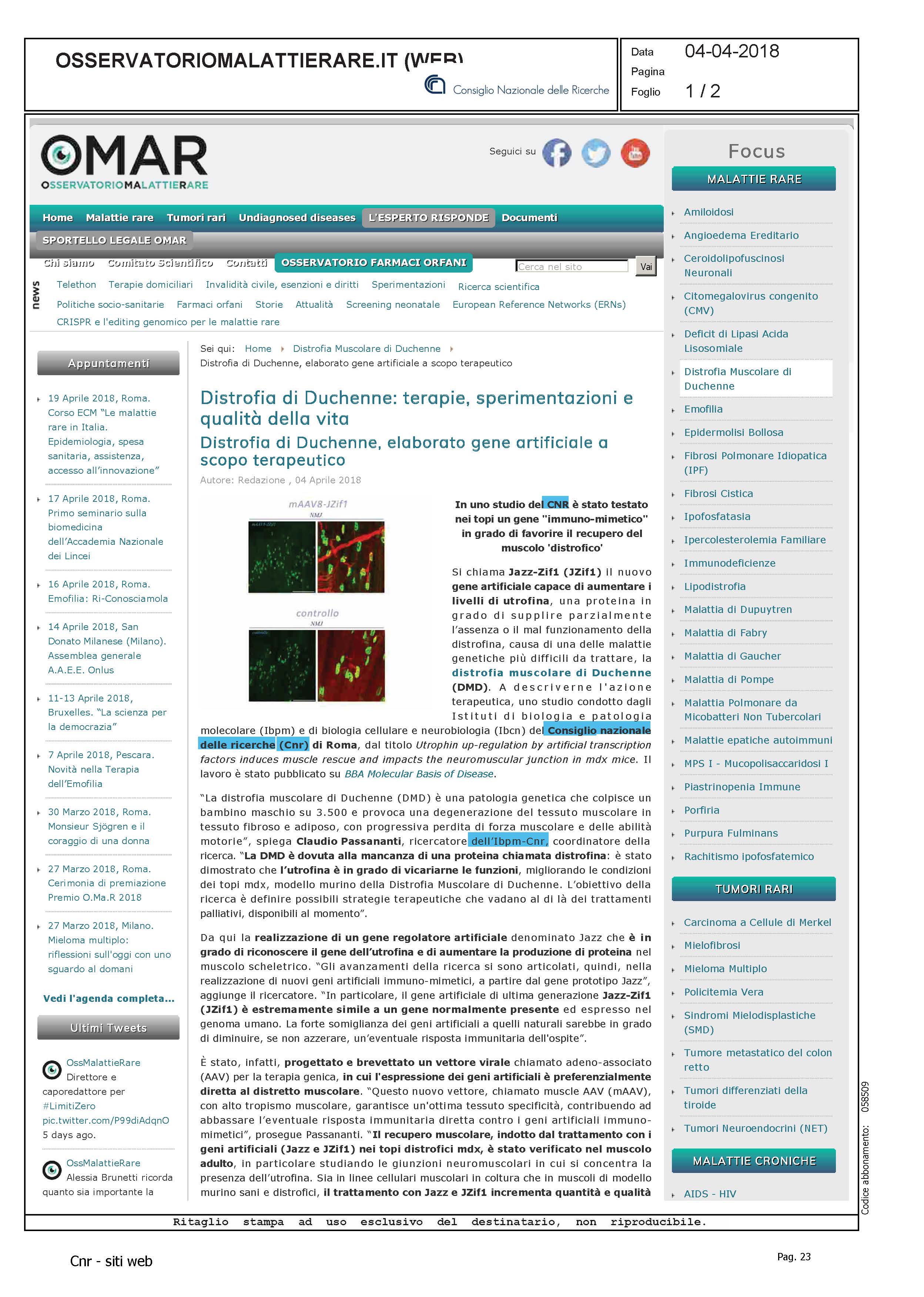
- “Geni artificiali come strategia terapeutica per la Distrofia muscolare di Duchenne” CLAUDIO PASSANANTI
- “DNA-GEL” UO-IBPM: PATRIZIA FILETICI (progetto coordinato da Sapienza)
Sarà inoltre presentato il nuovo bando per le infrastrutture di ricerca.

| DNA-GEL |
MITO-IMAGING |
PREMIO A FEDERICA POLVERINO PER IL MIGLIOR POSTER NEL CONVEGNO CONGIUNTO PHD ABCD-SIBBM 2018
Documenti: National Ph.D. Meeting Award
Highlights
09-20-18Giornata di presentazione della rete di Biologia Strutturale del CNR (SBN@CNR), Roma, 16 Aprile 2025
Consiglio Nazionale delle Ricerche - Aula Marconi
Piazzale Aldo Moro 7 - Roma
Cari colleghi e care colleghe,
Il convegno da noi organizzato ha lo scopo di creare un network di biologia strutturale (SBN-CNR) per potenziare l’impatto della biologia strutturale del CNR, perché possa essere di aiuto a tutta la comunità scientifica italiana nella risoluzione di problemi biologici e nella progettazione di nuovi “lead compound” attraverso lo “Structure Based Drug Design”. La diffusione di tecniche innovative come la “Cryo-EM” e la possibilità di predire le strutture di proteine sfruttando programmi di intelligenza artificiale come “AlphaFold”, hanno rivoluzionato la biologia strutturale. L'espansione delle conoscenze strutturali richiede nuovi approcci e una cooperazione interdisciplinare, missione centrale della rete SBN@CNR.
Speriamo di incontrarvi numerosi alla giornata.
Il comitato organizzatore
Link evento: https://sites.google.com/view/rete-sbn-cnr
Link programma: https://sites.google.com/view/rete-sbn-cnr/programma
Mostra "Vaccination, a time machine", Londra, luglio 2024



Arte e storia per divulgare l'immunologia
Francesca Di Rosa, dirigente di ricerca presso il Cnr-Ibpm e visiting scientist presso il Francis Crick Institute di Londra, ha curato un’originale mostra scientifica per l’edizione 2024 della prestigiosa Royal Society Summer Exhibition svolta lo scorso luglio nella capitale inglese. In occasione di questo evento divulgativo a cadenza annuale, la Royal Society apre le porte del suo edificio storico nel centro di Londra e accoglie migliaia di visitatori interessati agli ultimi progressi della scienza e della tecnologia in Gran Bretagna.
Titolo della mostra, realizzata in collaborazione con il prof. Adrian Hayday del Francis Crick Institute e King’s College London era “Vaccination, a time machine”. Il Francis Crick Institute era Istituzione Leader della mostra, mentre il Consiglio Nazionale delle Ricerche (CNR), il King’s College London (KCL), e il Centre of the Cell, Queen Mary University of London (CotC) Istituzioni Partner. Per suscitare curiosità e interesse nei visitatori, è stato scelto un approccio multidisciplinare, come la stessa ricercatrice ha raccontato nell’articolo pubblicato su Nature Review Immunology “Using art and history to communicate immunology to a broad audience”.
Il progetto ha riscosso molto successo: “L’obiettivo era quello di suscitare curiosità e allargare le conoscenze dei visitatori mettendo in relazione questioni scientifiche attuali con documenti storici provenienti dall'archivio della Royal Society, e opere d’arte contemporanea”, spiega Di Rosa “Ad esempio, il trittico di sculture di vetro “Passato, presente, futuro” dell’artista Luke Jerram, raffigurante il virus del vaiolo, il virus dell’immunodeficienza umana (HIV) e un virus immaginario originato da un’ipotetica mutazione, è stato messo in mostra per avvicinare in maniera inusuale i visitatori alla tematica dell’evoluzione virale, e a quella dei meccanismi di difesa contro vecchi e nuovi agenti infettivi. Per la parte storica, sono stati esposti antichi testi sulla variolizzazione, una misura preventiva che rappresenta l’antenato della moderna vaccinazione. Clessidre e giochi interattivi illustrati da giovani animatori scientifici sono stati impiegati per simulare l'impatto del tempo sull'immunità. Così, alcune clessidre progettate ad hoc sono state usate per spiegare la protezione immunitaria in diversi momenti post-vaccinazione, mentre giochi interattivi sono stati l’occasione per discutere il tema della cosiddetta immunità di gregge. Una coreografia creata appositamente dalla Royal Academy of Dance è stata il mezzo per evocare in maniera accattivante i meccanismi della vaccinazione”.
Il formato della mostra, a cavallo tra scienza, storia e arte, è in accordo con il principio delle "5C" formulato nell’articolo, capisaldi che, secondo la ricercatrice, hanno orientato la progettazione e la realizzazione di questo innovativo progetto di comunicazione scientifica: Concetto della mostra, incentrato sul contenuto scientifico da declinare attraverso più modalità; Collaborazione interdisciplinare; Coerenza nel messaggio; Conversazione aperta tra i membri del team e il pubblico; Continuità dell'esperienza, che non si conclude con la chiusura della mostra ma prosegue attraverso scambi personali, messaggi email, canali social, podcast.
Per informazioni:
Francesca Di Rosa
Cnr-Ibpm
francesca.dirosa@cnr.it
3/10/2024
IBPM-CNR @ the European Biotech Week 2024 (EBW2024)
"CACCIA AL DNA: INVESTIGANDO SULLA SCENA DEL CRIMINE"
Giovedì 3 Ottobre , 10 :00-14:00 - Sede Centrale CNR - Aula Marconi, piazzale Aldo Moro 7, Roma
Una giornata investigativa per le classi delle scuole secondarie che offre un'esperienza simulata del “DNA profiling” utilizzato nei laboratori forensi. Un tour virtuale guiderà gli studenti attraverso le fasi chiave della comprensione del genoma.

Organizzazione evento IBPM-CNR per la EBW2024: Teresa Colombo, Giovanna Costanzo, Alessandra Guidi
Link: https://www.cnr.it/it/evento/19381
Link: https://assobiotec.federchimica.it/agenda/tutti-gli-eventi/2024/09/24/default-calendar/biotech-week-2024
27/09/2024
IBPM-CNR @ the European Researchers' Night 2024 (ERN2024)
EVENTO:
"DNA take away"
27 Settembre - 28 Settembre 2024, 18:30-23:00 - Città dell'Altra Economia, Testaccio, Roma
Largo Dino Frisullo, snc, 00153 Roma RM
Evento ad accesso gratuito, dagli 8 anni. Attenzione: è necessaria la prenotazione online sul sito di https://www.scienzainsieme.it/ (La pagina diretta per l'evento DNA Take Away è: https://www.scienzainsieme.it/dna_take-away/) per partecipare all'attività sperimentale
Un esperimento da scienziati! I partecipanti saranno coinvolti in uno speciale esperimento, semplice ma scientificamente rigoroso, che permetterà di estrarre il proprio DNA e realizzarne un “fiocco” da mettere in una bottiglietta. Cosa farci poi? Portarlo con sé, per ricordarsi quanto la scienza sia sorprendente!

Link: https://www.scienzainsieme.it/notte-europea-dei-ricercatori/
22/11/2023
Nel DNA della popolazione nomade dei Fulani la testimonianza di antichi pastori sahariani
Uno studio, condotto da un team internazionale coordinato da Sapienza Università di Roma e dall’Istituto di biologia e patologia molecolari del Consiglio nazionale delle ricerche, mostra un’affinità genetica tra Fulani e individui del Marocco risalenti al Neolitico. I risultati, pubblicati sulla rivista Current Biology, individuano nei pastori nomadi Fulani i discendenti di una antica po-polazione “pan-Sahariana” che si è poi definitivamente frammentata e divisa con la desertificazio-ne della regione africana
Eugenia D'Atanasio eugenia.datanasio@cnr.it
Fulvio Cruciani fulvio.cruciani@uniroma1.it
Un team di ricerca internazionale, coordinato dalla Sapienza e dall’Istituto di biologia e patologia molecolari del Consiglio nazionale delle ricerche (Cnr-Ibpm) di Roma, ha condotto uno studio che fa luce sul ruolo dei cambiamenti climatici del Sahara.
Sfruttando l’informazione contenuta nell’intero genoma di 43 individui e applicando le nuove tecnologie di sequenziamento ad alta copertura, i ricercatori si sono focalizzati sui Fulani, la più estesa popolazione di pastori nomadi al mondo, attualmente stanziata lungo tutta la fascia saheliana, immediatamente a sud del deserto del Sahara. Questa popolazione, da studi genetici e antropologici precedenti, sembrava presentare delle caratteristiche peculiari, diverse da quelle delle popolazioni circostanti.
Lo studio, pubblicato sulla rivista Current Biology, ha indagato la natura di questa specificità al fine di darne anche una caratterizzazione storica e cronologica attraverso il confronto con altri genomi moderni e antichi presenti in letteratura.
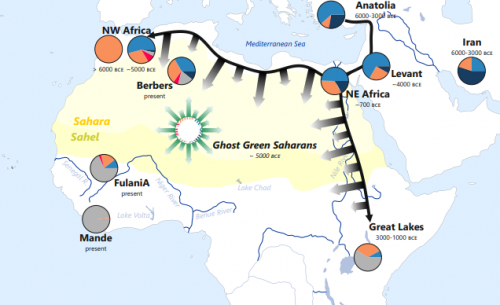
Rappresentazione semplificata delle componenti genetiche nelle popolazioni del Green Sahara e nei Fulani.
(Credits: Eugenia D'Atanasio. Concessione di Current Biology)
Per la prima volta è stata evidenziata una forte affinità genetica, finora solo ipotizzata sulla base di elementi culturali e reperti archeologici, tra i Fulani e individui del Marocco risalenti al Neolitico. Con un approccio di analisi congiunta di genomi moderni e antichi mai applicato per il continente africano, è stato possibile collocare l’origine dei Fulani nell’arco dell’ultimo periodo umido del Sahara (12.000 – 5.000 anni fa, denominato “Green Sahara”), quando l’attuale deserto era invece una terra fertile, rigogliosa e abitata, facendo di questa popolazione la testimone chiave di uno dei più grandi cambiamenti climatici del continente africano.
“Gli approcci utilizzati in questo studio ci hanno inoltre permesso di fare luce sulla storia recente dei Fulani, che risultano suddivisi in due sotto-gruppi geneticamente distinti a causa di fattori socio-culturali non ancora ben caratterizzati”, spiega Flavia Risi, dottoranda in genetica e biologia molecolare presso la Sapienza Università di Roma e co-autrice della ricerca.
Lo scenario ipotizzato sulla base di questi risultati propone inoltre la presenza nel “Green Sahara” di frequenti contatti e migrazioni tra le popolazioni, a formare una sorta di popolazione “pan-Sahariana” che si è poi definitivamente frammentata e divisa con la desertificazione dell’area, a partire da 5.000 anni fa ma di cui rimane traccia nei Fulani in virtù di uno stile di vita particolarmente isolato.
“L’ambiente fertile del Sahara ha sicuramente promosso estesi movimenti e contatti tra le popolazioni del Sahara e i risultati di questo studio aiutano a tracciare una linea di congiunzione tra gli antichi pastori sahariani e i moderni Fulani” spiega Eugenia D’Atanasio, ricercatrice dell’Istituto di biologia e patologia molecolari del Cnr (Cnr-Ibpm) e co-coordinatrice dello studio.
Lo studio rappresenta un contributo alle conoscenze attuali sulla storia evolutiva umana e sulla sua biodiversità, in particolare nel continente africano, luogo che ospita la più grande ricchezza genetica per la nostra specie. Tali risultati evidenziano poi come spostamenti e contatti transahariani non siano solo un fenomeno recente, ma una realtà storica antichissima, confermando infine il ruolo dei grandi cambiamenti climatici (come la desertificazione) nelle migrazioni umane.
“L’Africa, nonostante la sua ricchezza culturale e genetica, è ancora poco studiata a livello genomico. Oltre alle informazioni sul popolamento del “Green Sahara”, le sequenze genomiche prodotte durante questo studio rappresentano uno strumento essenziale per la comunità scientifica in quanto permettono di colmare, almeno in parte, una grossa lacuna nella nostra conoscenza della diversità umana”, aggiunge Fulvio Cruciani del Dipartimento di Biologia e biotecnologie Charles Darwin della Sapienza Università di Roma e PI del progetto.
18/09/2023
IBPM-CNR @ the European Researchers' Night 2023 (ERN2023)
EVENTO:
"DNA take away"
29-30 Settembre 2023, 18:30-23:00 - Città dell'Altra Economia, Testaccio, Roma
Evento ad accesso gratuito, ma è consigliata la PRENOTAZIONE
Un esperimento da scienziati! I partecipanti saranno coinvolti in uno speciale esperimento, semplice ma scientificamente rigoroso, che permetterà di estrarre il proprio DNA e realizzarne un “fiocco” da mettere in una bottiglietta. Cosa farci poi? Portarlo con sé, per ricordarsi quanto la scienza sia sorprendente!

Link: https://www.scienzainsieme.it/notte-europea-dei-ricercatori/
18/09/2023
IBPM-CNR @ the European Biotech Week 2023 (EBW2023)
EVENTO 1:
"A spasso nel tuo genoma"
Giovedì 28 Settembre, 9:00-14:00 - Sede Centrale CNR - Aula Marconi, piazzale Aldo Moro 7, Roma
Una giornata di esplorazione del nostro genoma dedicata agli studenti della scuola secondaria superiore. Racconteremo le tappe fondamentali della conoscenza del genoma umano e come questa conquista abbia cambiato il volto della ricerca bio-medica, la visione che abbiamo dell’organizzazione del nostro genoma e la comprensione dei meccanismi alla base di molte malattie. Compiremo un tour virtuale del genoma umano utilizzando una sorta di Google Maps genomico. Non mancheranno quiz per esplorare numeri e fatti sorprendenti sul nostro genoma. Tra una presentazione e una tappa del nostro tour, i partecipanti potranno prendere parte ad una vera e propria estrazione del proprio DNA. Non solo, per la prima volta in questa edizione i ragazzi potranno contare sul supporto di un assistente d’eccezione durante il laboratorio: un “edudroide”, ovvero un robot interattivo pronto a risolvere dubbi e curiosità sul DNA.
EVENTO 2:
"Un tuffo nel magico mondo dell’acqua!"
26 Settembre 2023 (9:00-13:00); 27 Settembre 2023 (9-16) - Scuola primaria dell'Istituto Comprensivo "Martin Luther King", via degli Orafi 30, Roma
L’acqua rappresenta da sempre per i bambini un mondo affascinante e di gran divertimento. Questo laboratorio si propone di esplorare le straordinarie proprietà dell’acqua, che sono ogni giorno sotto i nostri occhi e che verranno spiegate ai bambini in maniera divertente e allo stesso tempo scientificamente rigorosa. Il laboratorio prevede di esplorare insieme alcune proprietà dell’acqua (temperatura, stato, capillarità, tensione superficiale e densità) attraverso un linguaggio semplice e con esperimenti mirati alla comprensione di tali caratteristiche.

Organizzazione evento IBPM-CNR per la EBW2023: Giovanna Costanzo, Teresa Colombo, Alessandra Guidi, Cecilia Mannironi, Erica Salvati
Link: https://www.cnr.it/it/evento/18667/il-cnr-ibpm-per-la-biotech-week-2023
Link: https://assobiotec.federchimica.it/agenda/tutti-gli-eventi/2023/09/25/default-calendar/biotech-week-2023
16/02/2023
Sharing the future - GIOVANI e SCIENZA
100 anni di Consiglio Nazionale delle Ricerche, che dal 1923 realizza progetti di ricerca scientifica nei principali settori della conoscenza per lo sviluppo del Paese, promuovendo l'innovazione, lʼinternazionalizzazione del "sistema ricerca" e favorendo la competitività del sistema industriale. Per celebrare questa importante ricorrenza, si aprono le porte agli studenti di diverse scuole secondarie di II grado di Roma e del Lazio.
Assoluti protagonisti dellʼincontro, organizzato da CNR - Unità Comunicazione insieme a Fondazione Mondo Digitale, ente del Terzo settore impegnato da oltre 20 anni nella diffusione della conoscenza e della cultura dell'innovazione, sono proprio le ragazze e i ragazzi, che hanno la possibilità di entrare in contatto con le ricercatrici e i ricercatori del CNR per approfondire progressi e scoperte su temi quali la salute dellʼuomo e del pianeta, ambiente ed energia, intelligenza artificiale.
Le sessioni, in cui si affrontano discussioni su robotica, scienze della vita, sostenibilità, energia pulita, intelligenza artificiale e transizione digitale, sono organizzate sul format del role modeling, per creare opportunità di confronto e scambio tra studenti e ricercatori. Lo scopo è quello di mostrare modelli di riferimento positivi per trasmettere valori e interessi che fungano da ispiratori per gli studenti partecipanti.

Organizzazione evento:
CNR Unità comunicazione
Fondazione Mondo DIgitale
Link pagina CNR: https://www.cnr.it/it/evento/18381/sharing-the-future-giovani-e-scienza
Link programma: Agenda_Sharing_the_future_CNR_sede_16febb23.pdf
19/09/2022
IBPM-CNR @ the European Biotech Week 2022 (EBW2022)
EVENTO 1:
"A spasso nel tuo genoma"
Lunedì 26 Settembre, 9:00-13:00 - Sede Centrale CNR - Aula Marconi, piazzale Aldo Moro 7, Roma
Una giornata di esplorazione del nostro genoma con gli studenti delle Liceo Ginnasio "T. Tasso" di Roma. Racconteremo le tappe fondamentali della conoscenza del genoma umano e come questa conquista abbia cambiato il volto della ricerca bio-medica, la visione che abbiamo dell’organizzazione del nostro genoma e la comprensione dei meccanismi alla base di molte malattie. Compiremo un tour virtuale del genoma umano utilizzando una sorta di Google Maps genomico. Non mancheranno quiz per esplorare numeri e fatti sorprendenti sul nostro genoma. Tra una presentazione e una tappa del nostro tour, i partecipanti potranno prendere parte ad una vera e propria estrazione del proprio DNA.
EVENTO 2:
"Un tuffo nel magico mondo dell’acqua!"
27-28-29-30 Settembre 2022, 9:00-12:00 - Scuola primaria ed infanzia "Vittorio Piccinini", Istituto Comprensivo "Anna Fraentzel Celli", via F. Fiorentini 48, Roma
L’acqua rappresenta da sempre per i bambini un mondo affascinante e di gran divertimento. Questo laboratorio si propone di esplorare le straordinarie proprietà dell’acqua, che sono ogni giorno sotto i nostri occhi, e che verranno spiegate ai bambini in maniera giocosa ma allo stesso tempo scientifica. Il laboratorio prevede di esplorare insieme alcune comuni proprietà dell’acqua (temperatura, stato, capillarità, tensione superficiale e densità) attraverso un linguaggio semplice e con esperimenti mirati alla comprensione di tali caratteristiche. L’attività viene proposta a due fasce di età (3-6 e 6-10) per le quali verranno affrontati i medesimi temi ma con modalità e linguaggio adattati alla fascia d’età dei partecipanti.

Organizzazione evento IBPM-CNR per la EBW2022: Giovanna Costanzo, Angela Cirigliano, Teresa Colombo, Alessandra Guidi, Cecilia Mannironi, Erica Salvati
Link: https://www.cnr.it/it/evento/18077/il-cnr-ibpm-per-la-biotech-week-2022
Link: https://assobiotec.federchimica.it/agenda/tutti-gli-eventi/2022/09/26/default-calendar/biotech-week-2022
19/09/2022
IBPM-CNR @ the European Researchers' Night 2022 (ERN2022)
EVENTO:
"Il mio DNA? Oggi lo metto in bottiglia"
30 Settembre - 01 Ottobre 2022, 18:30-23:00 - Città dell'Altra Economia, Testaccio, Roma
Evento ad accesso gratuito, ma è consigliata la PRENOTAZIONE
Un esperimento da scienziati! I partecipanti saranno coinvolti in uno speciale esperimento, semplice ma scientificamente rigoroso, che permetterà di estrarre il proprio DNA e realizzarne un “fiocco” da mettere in una bottiglietta. Cosa farci poi? Portarlo con sé, per ricordarsi quanto la scienza sia sorprendente!

Link: https://www.scienzainsieme.it/il-mio-dna-oggi-lo-metto-in-bottiglia/
Link: https://www.scienzainsieme.it/category/notte-2022/roma/
20/06/2022
La prima sequenza completa del genoma umano: IBPM ne parla all'Italia con Voi
Lo scorso 1 Aprile 2022 è stato pubblicato sulla rivista Science il lavoro del consorzio Telomere-2-Telomere (T2T) che illustra un nuovo traguardo fondamentale per la biomedicina: la prima sequenza completa, da telomero-a-telomero, di un genoma umano. Due ricercatrici dell'IBPM, Teresa Colombo e Giovanna Costanzo, ne parlano alla trasmissione "L'Italia con Voi" del canale RAI Italia andata in onda lo scorso 14 Giugno e accessibile sul sito RaiPlay a questo link: https://www.raiplay.it/video/2022/06/LItalia-con-Voi-Puntata-del-14062022-16384347-0525-4f70-a9dc-20c25f2e9035.html (intervento IBPM intorno al minuto 45')
01/02/2022
Treatment of kidney clear cell carcinoma, lung adenocarcinoma and glioblastoma cell lines with hydrogels made of DNA nanostars
Biomaterials Science
Link: https://pubs.rsc.org/en/content/articlelanding/2022/bm/d1bm01643a
DNA-GEL, nuovi nanomateriali composti da stelline di DNA, da utilizzare nel trattamento di linee cellulari tumorali.
10/01/2022
AVVISO DI DISPONIBILITA’ PER UN ASSEGNO DI RICERCA CNR
Sarà pubblicato a breve il bando per un assegno di ricerca presso l’IBPM, sede di via degli Apuli 4. L’assegno di ricerca di 1 anno, rinnovabile, è bandito nell’ambito del programma MUR “PNIR - Programma Nazionale Infrastrutture di Ricerca” per il rafforzamento del capitale umano delle Infrastrutture di Ricerca.
In particolare l’AdR sarà dedicato al rafforzamento del personale nell’ambito del progetto PON “IMPARA – Imaging dalle molecole alla preclinica”, progetto Nazionale guidato dal CNR per lo sviluppo dell’infrastruttura nazionale di Imaging
http://www.ponricerca.gov.it/comunicazione/esempi-di-progetto/potenziamento-infrastrutture-di-ricerca/impara-imaging-dalle-molecole-alla-preclinica/
a sua volta dedicato all’ampliamento dell’infrastruttura Europea di imaging Eurobioimaging
https://www.eurobioimaging.eu/
Nell’ambito del progetto IMPARA, l’AdR collaborerà alla realizzazione del seguente obiettivo:
Sviluppo di metodologie di imaging per lo sviluppo di strategie genetiche e farmacologiche di potenziale valore terapeutico.
Qualificazioni, competenze, profilo scientifico e titoli di studio richiesti
Titoli di studio: Laurea Magistrale in Biologia (LM-06) o Biotecnologie (LM-09)
Esperienza in: colture cellulari di mammifero; microscopia in campo chiaro e fluorescenza per l’analisi di campioni fissati e vitali (video-registrazione).
I candidati interessati sono pregati di prendere contatto con
Patrizia Lavia patrizia.lavia@uniroma1.it
17/09/2021
POR FESR 2014-2020 Avviso Pubblico “Gruppi di Ricerca - 2020” Progetti finanziati

APTASER - Sviluppo di inibitori ad acido nucleico della serina idrossimetiltrasferasi nel tumore al polmone
DWARF - La felce Pteris vittata: una strategia green per eliminare l’arsenico dalle acque potabili
INNOVA3DIMAGING - Metodologie innovative di 3D high content imaging per la validazione di nuove molecole antitumorali
NEWPAT - Analisi genetica del DNA fetale circolante nel sangue materno: sviluppo di un test di paternità non invasivo ad elevata specificità
ORGANOVA - Sviluppo di modelli organotipici e organ-on-chip di tumore ovarico per testare farmaci antitumorali
TOC - interazioni tra luce e ormoni nel controllo dell’apertura fiorale in specie modello e ortive
SMART-BREED - Tecnologie molecolari innovative per l’adattamento delle specie ortive al cambiamento climatico mediante breeding di precisione
27/07/2021
Coordination of biradial-to-radial symmetry and tissue polarity by HD-ZIP II proteins
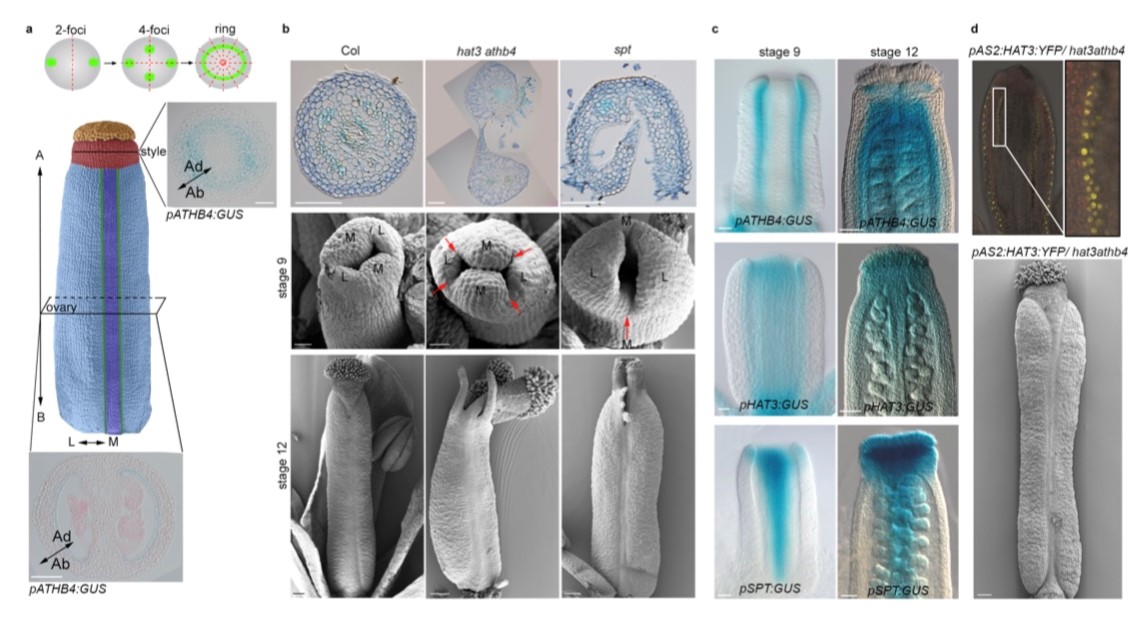
L'organo riproduttivo femminile, il gineceo, della pianta modello Arabidopsis thaliana è finora l'unico esempio noto studiato a livello molecolare di una struttura in via di sviluppo che si riprogramma nel tempo per ottenere una transizione da simmetria bilaterale a radiale.
In uno studio condotto presso l’IBPM-CNR in collaborazione con il John Innes Center (UK) e il CREA-GB (Roma) è stato studiato il ruolo dei fattori di trascrizione HAT3 e ATHB4 nel passaggio da simmetria bilaterale a simmetria radiale durante la formazione dello stilo, la parte apicale del gineceo, nella pianta modello Arabidopsis thaliana. In precedenti studi, era stato dimostrato che il cambiamento da simmetria bilaterale a radiale del gineceo avviene attraverso vari stadi che possono essere descritti da un cambiamento della distribuzione dell’ormone auxina. In particolare, nella parte apicale del gineceo in formazione, l’auxina passa da una distribuzione a due foci (simmetria bilaterale) ad una a 4 foci (biradiale), ed infine ad un anello completo (radiale). Questo processo è controllato in modo cooperativo dai fattori di trascrizione bHLH SPATULA (SPT), INDEHISCENT e HECATE1, 2 e 3. I relativi mutanti, spatula e indehiscent sono bloccati allo stadio bilaterale a due foci, mentre il mutante triplo hecate1, 2, 3 è bloccato allo stadio di quattro foci. Nel lavoro appena pubblicato sulla rivista Nature Communications, viene descritto il ruolo di HAT3 e ATHB4, due fattori di trascrizione noti come target diretti di SPATULA, per il completamento del processo che porterà alla formazione dell’organo radializzato. Nel lavoro viene mostrato che i geni HAT3 e ATHB4 sono controllati positivamente anche dai geni HECATE in modo cooperativo con SPATULA. Attraverso analisi genetiche, l’utilizzo di fattori di trascrizione inducibili e di marcatori fluorescenti si è riusciti a stabilire che HAT3 e ATHB4 controllano la distribuzione dell’auxina e la sensibilità all’ormone citochinina. E’ stato inoltre osservato un controllo a feedback negativo di HAT3 e ATHB4 sul gene SPATULA. Questa regolazione è molto probabilmente responsabile del corretto bilancio della segnalazione ormonale nel tempo e nello spazio di auxina e citochinina, in modo da permettere la transizione della simmetria e la formazione del corretto organo radializzato.
Con questo lavoro, si vuole ricordare la collega Ida Ruberti, purtroppo venuta a mancare durante la fase finale di questo lavoro, per il suo ruolo fondamentale nella concettualizzazione del progetto e le approfondite discussioni sui risultati.

07/07/2021
Aspettando la Notte Europea Dei Ricercatori Talenti per la Scienza - 7, 9 E 10 Luglio 2021

Documenti:
NET_TALENTI-PER-LA-SCIENZA.pdf
Mappa TALENTI-.pdf
06/07/2021
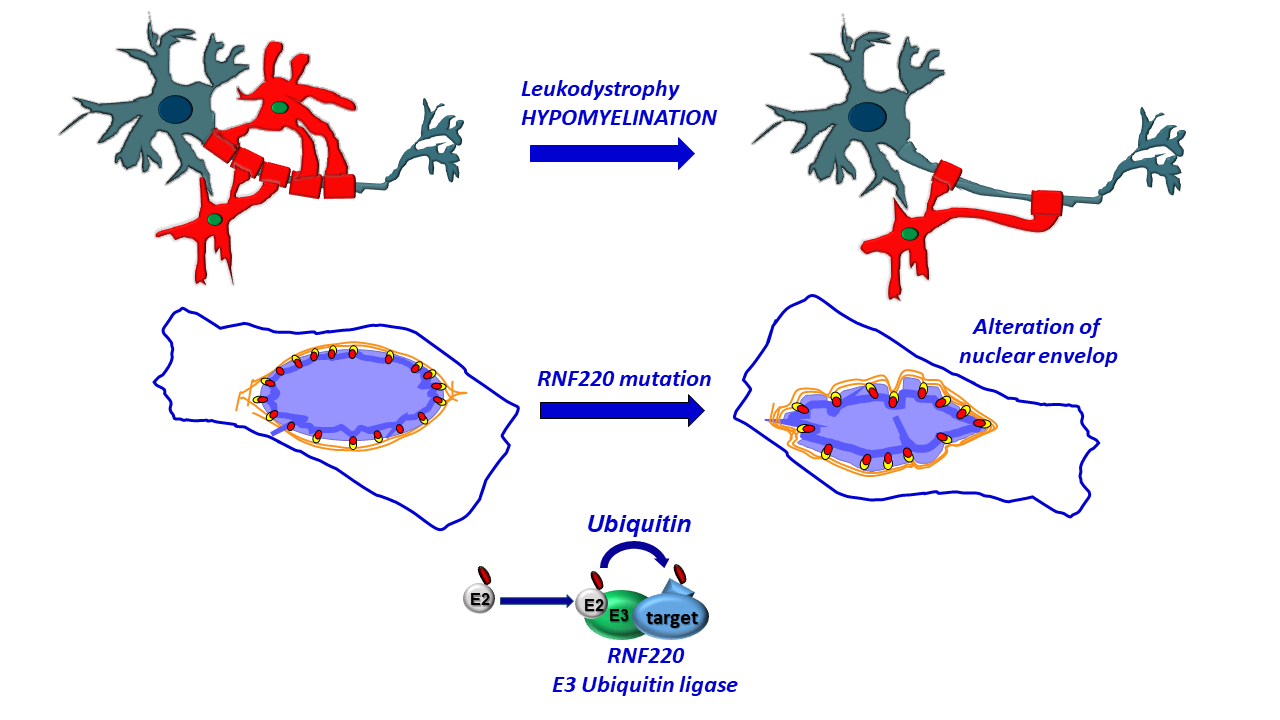
Biallelic mutations in RNF220 cause laminopathies featuring leukodystrophy, ataxia and deafness
Impieghiamo la Drosophila melanogaster per scoprire i meccanismi molecolari alla base dei disturbi neurologici e neurodegenerativi, e stiamo studiando una nuova forma di leucodistrofia, legata all'atassia e alla sordità neurosensoriale.
Il sequenziamento dell'intero esoma di diverse grandi famiglie consanguinee, eseguito dai nostri collaboratori, ha permesso di identificare due mutazioni missenso che colpiscono residui conservati dell'ubiquitina E3 ligasi RNF220, come causative di questa forma di leucodistrofia. L'analisi di spettrometria di massa ha identificato la lamina B1 come partner di legame di RNF220. Abbiamo dimostrato che il silenziamento di RNF220 in Drosophila influisce in modo specifico sulla localizzazione subcellulare della lamina B1 e ne promuove l'aggregazione. Inoltre, la perdita di funzione di RNF220 promuove una forte neurodegenerazione nei tessuti del moscerino, supportando un legame funzionale tra RNF220 e la lamina B1.
Pertanto, dimostriamo che RNF220 è implicato in una nuova forma di leucodistrofia caratterizzata da atassia e ipoacusia neurosensoriale e mostriamo il suo ruolo nella regolazione della lamina nucleare. Inoltre, stabiliamo un modello di malattia in moscerino che potrebbe essere sfruttato per progettare nuove strategie terapeutiche.
03/06/2021
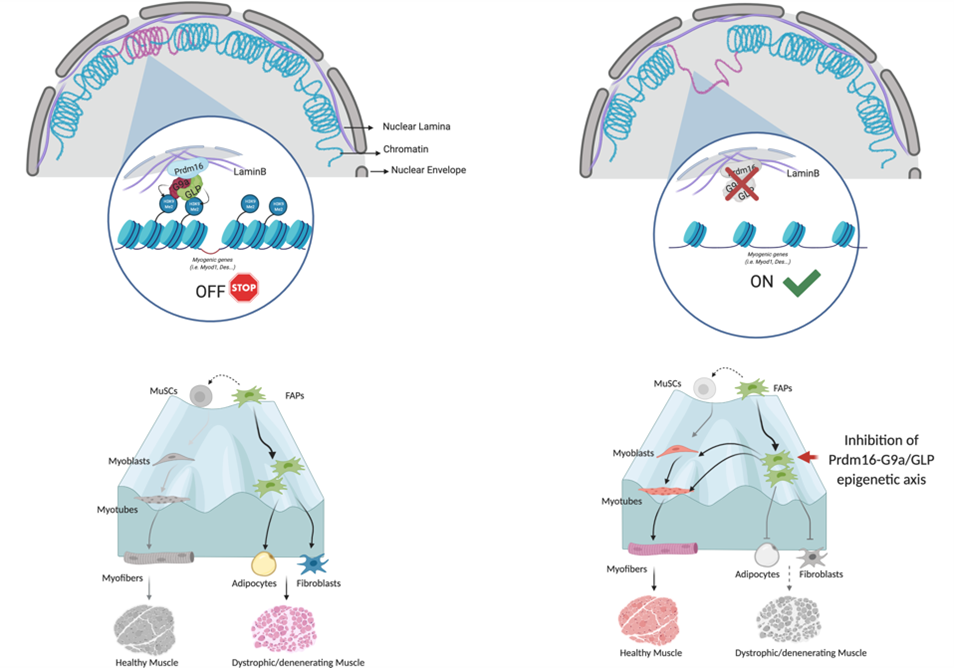
Prdm16-mediated H3K9 methylation controls fibro-adipogenic progenitors identity during skeletal muscle repair
Biferali et. al. (DOI: 10.1126/sciadv.abd9371)
In questo lavoro è stata studiata una popolazione di cellule stromali (i progenitori fibro-adipogenici, FAPs) che svolge un ruolo chiave nel mantenimento dell’omeostasi muscolare e della rigenerazione del muscolo scheletrico. I FAPs hanno, infatti, un duplice ruolo, da un lato promuovono la rigenerazione muscolare e dall'altro contribuiscono direttamente alla degenerazione fibro-adipogenica dei muscoli malati, quali ad esempio quelli dei pazienti affetti dalla distrofia muscolare di Duchenne.
In questo studio è stato individuato un meccanismo molecolare di repressione genica svolto dalle proteine Prdm16/G9a/GLP che è cruciale nel mantenere silente la capacità dei FAP di promuovere la formazione di tessuto muscolare. In particolare, questo lavoro mostra che la proteina Prdm16 gioca un ruolo fondamentale nel bloccare le regioni di DNA codificanti il potenziale muscolare dei FAP alla periferia nucleare, reclutando su di esse gli enzimi G9a e GLP per mantenerle silenti. Questo scoperta ha un importante risvolto traslazione e potenziale terapeutico, poiché è stato dimostrato che inibendo queste proteine in modelli murini di rigenerazione e distrofia, si riesce a cambiare il destino di queste cellule promuovendo la rigenerazione dei muscoli distrofici e allo stesso tempo rallentandone la degenerazione.
Referente IBPM: Chiara Mozzetta
Link: https://advances.sciencemag.org/content/7/23/eabd9371

04/03/2021
Cell Reports - Endothelin-1 drives invadopodia and interaction with mesothelial cells through ILK
Questo studio mette in luce come l’interazione molecolare tra le proteine ILK e ?-arrestina 1, a valle del recettore A dell’endotelina (ETAR) rappresenti un prerequisito per sostenere il processo metastatico delle cellule del carcinoma ovarico mediato dagli invadopodi. È stato chiarito che il complesso multiproteico contenente ILK e ?-arrestina 1 controlla dei circuiti di segnale finemente regolati e convergenti sulla GTPasi Rac3. L’attivazione specifica di questi segnali facilita la creazione di interazioni produttive tra cellule di carcinoma ovarico e cellule mesoteliali, e permette alle cellule tumorali di rimodellare la matrice extracellulare e invadere.
L’efficacia di Ambrisentan, un farmaco che controlla selettivamente l’attività di ETAR, nell'inibire l'adesione delle cellule tumorali ai siti metastatici e la loro capacità di formare metastasi in modelli preclinici, suggerisce che il controllo di questi circuiti di segnale attraverso il blocco di ETAR potrebbe rappresentare una via nel trattamento di questo tumore in fase pre-metastatica.
Lo studio è realizzato con il sostegno di AIRC ed hanno collaborato ricercatori dell’Istituto Regina Elena di Roma, Istituto Superiore di Sanità e Campus Biomedico di Roma.
DOI:https://doi.org/10.1016/j.celrep.2021.108800
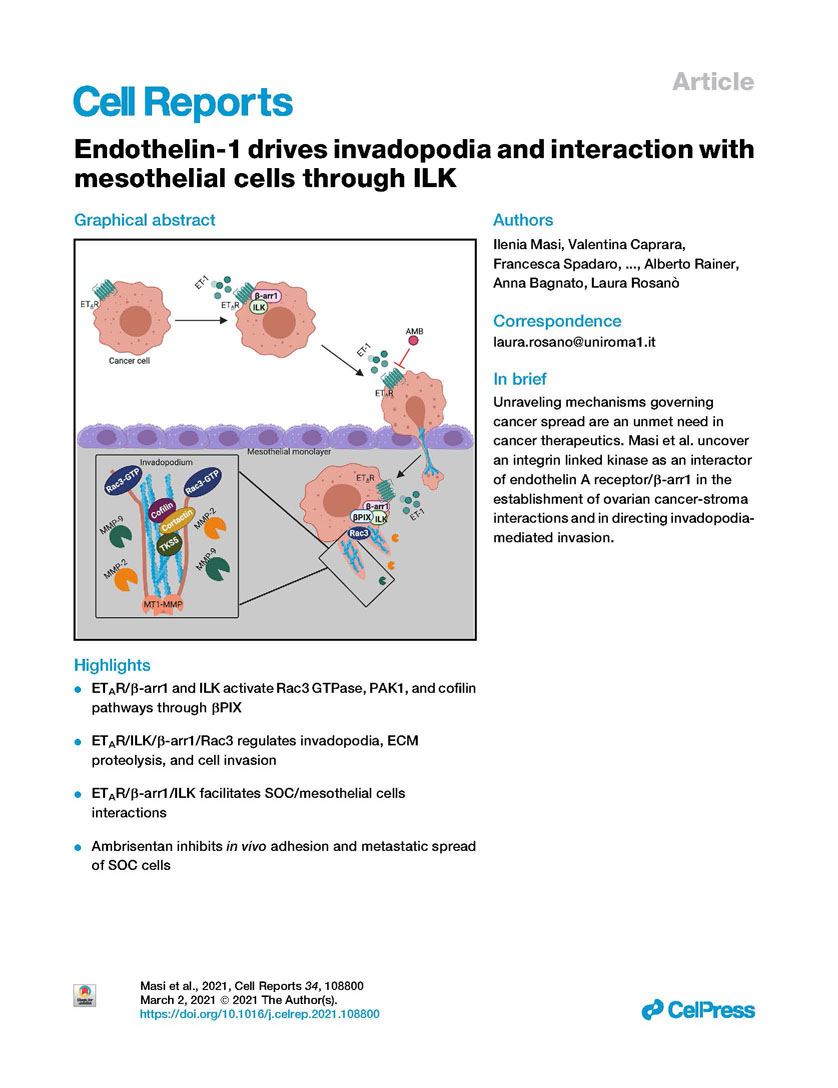
Documents: Endothelin-1 drives invadopodia and interaction with mesothelial cells through ILK.pdf
01/02/2021
Una "Green Revolution" del Cnr tra i vincitori della StartCup Lazio 2020
Tra i vincitori della Start Cup Lazio 2020 c’è il team CNR composto dalla Dr Patrizia Brunetti (IBPM-CNR), dal Dr Davide Marzi (IBPM-CNR) e dal Dr Giancarlo Daniele (EGATO-VT1), che con il progetto Hydro Fern ottiene la Menzione Speciale “Social Innovation”, il Premio Speciale Regione Lazio, si aggiudica il 4° posto ed ottiene l’accesso al Premio Nazionale dell’Innovazione (PNI 2020).

Documents: 5_Green-Clean-Biotech_LAZIO7_Avvenire_24_01_2021_pag3.pdf
Links:
https://www.cnr.it/it/news/9768/una-green-revolution-del-cnr-tra-i-vincitori-della-startcup-lazio-2020
https://www.tg24.info/regione-start-cup-lazio-2020-i-vincitori-della-6a-edizione/
https://www.repubblica.it/economia/rapporti/impresa-italia/mercati/2020/10/27/news/ecco_i_vincitori_della_6a_edizione_di_start_cup_lazio_2020-272047189/
28/01/2021
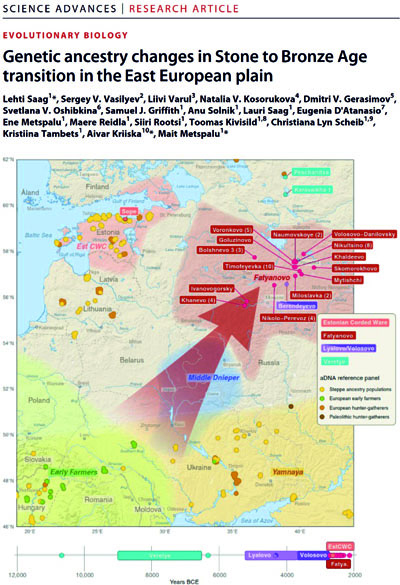
In questo studio, abbiamo prodotto sequenze dell’intero genoma da 30 individui antichi della Russia occidentale, più precisamente 3 cacciatori-raccoglitori dell’Età della Pietra (10800-4250 a.C.) e 26 agricoltori della Cultura di Fatyanovo (complesso della ceramica cordata, Età del Bronzo) (2900-2050 a.C.). L’analisi sul DNA antico ci ha permesso di mostrare che l’ancestry dei cacciatori-raccoglitori dell’Europa orientale era già presente in Russia dal 10000 a.C. Inoltre, con l’arrivo dell’agricoltura nell’area, si osserva un cambiamento nell’ancestry, con gli individui Fatyanovo che mostrano sia l’ancestry delle Steppe che quella dei primi agricoltori, in modo analogo ad altri gruppi della ceramica cordata. Infine, abbiamo potuto determinare il genotipo di 113 marcatori informativi per diverse caratteristiche fenotipiche, osservando cambiamenti nelle frequenze alleliche tra i cacciatori-raccoglitori e gli agricoltori Fatyanovo per marcatori coinvolti nella pigmentazione e nell’adattamento alla dieta. In conclusione, i nostri dati sembrano suggerire che le popolazioni associate alla cultura Fatyanovo probabilmente si sono originate in seguito ad una migrazione rapida da una regione vicina all’odierna Ucraina, dove le ancestry delle Steppe e dei primi agricoltori coesistevano già dal 3000 a.C. circa.
Referente IBPM: Eugenia D'Atanasio
15/12/2020

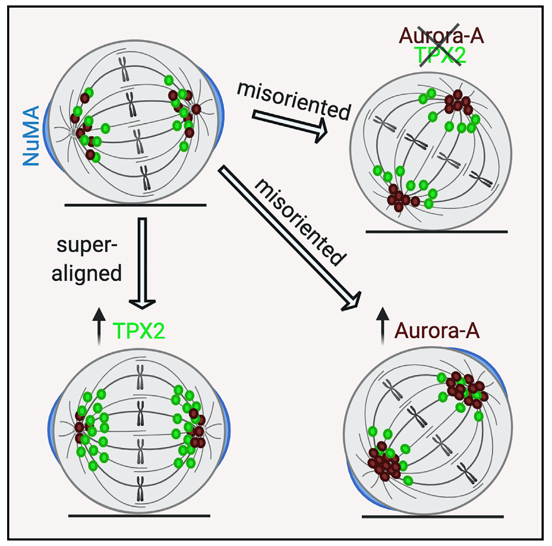 In questo studio è stato caratterizzato il ruolo della chinasi Aurora-A e del suo principale regolatore TPX2 nel controllo dell’orientamento del fuso mitotico in cellule di mammifero, processo che determina l’orientamento della divisione cellulare e di conseguenza il posizionamento ed il destino delle cellule figlie. Abbiamo messo in luce che TPX2 gioca un ruolo chiave nella regolazione da parte di Aurora-A della proteina del complesso di orientamento NuMA. Abbiamo anche descritto effetti opposti, dipendenti dai microtubuli, in seguito all’aumento dei livelli di Aurora-A o TPX2 in cellule non trasformate, risultanti rispettivamente in fusi “mis-orientati” o “super-allineati”. Poiché livelli elevati di Aurora-A e TPX2 sono spesso osservati nei tumori, lo studio fornisce una nuova chiave per comprendere i loro ruoli pro-tumorigenici. Apre anche la strada all’esplorazione della funzione di queste due proteine in nuovi contesti patologici, in particolare patologie dello sviluppo o neurologiche associate a difetti dell’orientamento della divisione cellulare.
In questo studio è stato caratterizzato il ruolo della chinasi Aurora-A e del suo principale regolatore TPX2 nel controllo dell’orientamento del fuso mitotico in cellule di mammifero, processo che determina l’orientamento della divisione cellulare e di conseguenza il posizionamento ed il destino delle cellule figlie. Abbiamo messo in luce che TPX2 gioca un ruolo chiave nella regolazione da parte di Aurora-A della proteina del complesso di orientamento NuMA. Abbiamo anche descritto effetti opposti, dipendenti dai microtubuli, in seguito all’aumento dei livelli di Aurora-A o TPX2 in cellule non trasformate, risultanti rispettivamente in fusi “mis-orientati” o “super-allineati”. Poiché livelli elevati di Aurora-A e TPX2 sono spesso osservati nei tumori, lo studio fornisce una nuova chiave per comprendere i loro ruoli pro-tumorigenici. Apre anche la strada all’esplorazione della funzione di queste due proteine in nuovi contesti patologici, in particolare patologie dello sviluppo o neurologiche associate a difetti dell’orientamento della divisione cellulare.
Lo studio è frutto di una collaborazione tra l’Istituto di Biologia e Patologia Molecolari (IBPM) e l’Istituto Europeo di Oncologia (IEO). Hanno partecipato gruppi di ricerca dell’Università Sapienza di Roma e del Max Planck Institute of Molecular Physiology (Dortmund, DE).
Referente IBPM: Giulia Guarguaglini
Comunicato CNR: https://www.cnr.it/it/comunicato-stampa/9861/nuova-luce-sulla-divisione-cellulare-nei-tumori
17/8/2020
Approfondimenti ai siti web:
https://rdcu.be/b6jAd
www.immunophenotype.org
twitter: immunosurveillance@TheCrick
Lo studio ha permesso di identificare nel sangue periferico dei pazienti con COVID-19 un insieme di cambiamenti a livello del sistema immunitario, che nell’insieme costituiscono un tratto caratterizzante questa malattia. È stato trovato che, specialmente nei pazienti più gravi, i linfociti T del sangue periferico sono tipicamente caratterizzati da una marcata linfopenia e da un fenotipo di attivazione/proliferazione. Alcuni dei cambiamenti identificati, ad esempio la notevole riduzione dei granulociti basofili e delle cellule dendritiche plasmacitoidi, correlano con la severità clinica della patologia. In particolare, è stato osservato che l’aumento dei livelli di una triade di molecole – IP-10, interleuchina-10 e interleuchina-6 – anticipa l’aggravamento della malattia nella coorte di pazienti studiata, informazione che potrà essere utile a scopi prognostici se confermata in un numero più elevato di pazienti.
Lo studio, denominato COVID-IP, è stato diretto dal prof. Adrian Hayday (Francis Crick Institute, King’s College London, Londra, Regno Unito). È stata studiata una coorte di 63 pazienti con COVID-19 ricoverati presso noti ospedali di Londra (Guy’s and St Thomas’ Hospitals). La dott.ssa Francesca Di Rosa dell’IBPM ha contribuito allo studio introducendo un saggio innovativo per l’analisi del ciclo cellulare dei linfociti T, usato qui per la prima volta in pazienti con COVID-19.
13/7/2020
L’identificazione della proteina FUS, coinvolta nell’insorgenza della Sclerosi Laterale Amiotrofica (SLA), quale interattore diretto di HOTAIRM1 in motoneuroni, insieme alla presenza di una isoforma citoplasmatica di HOTAIRM1 localizzata nel soma e nei neuriti (frecce gialle) dei motoneuroni, suggerisce un possibile coinvolgimento di questo trascritto non codificante in processi neurodegenerativi.
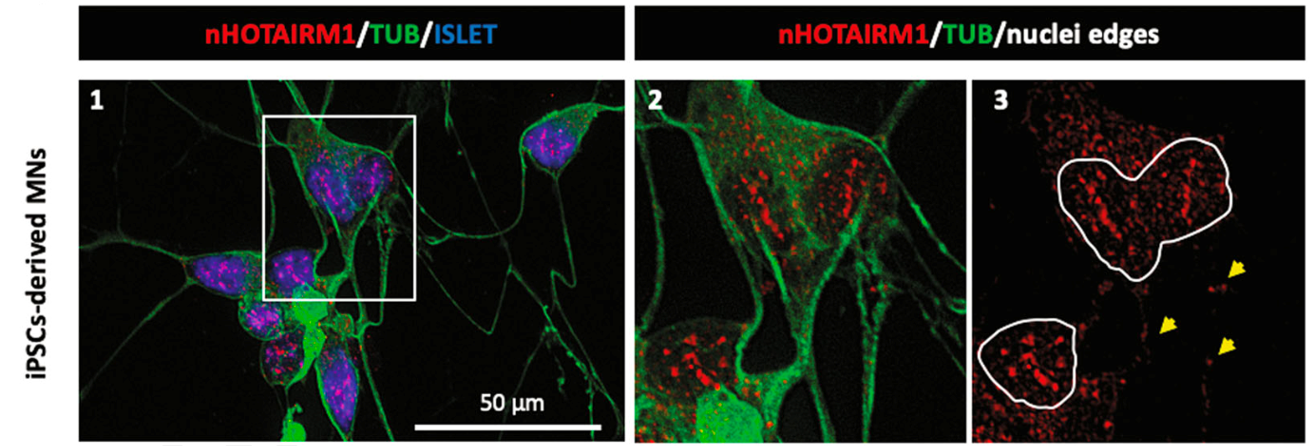
Lo studio è il frutto di una collaborazione tra Istituto di Biologia e Patologia Molecolari (IBPM-CNR), Dipartimento di Biologia e Biotecnologie (Sapienza Università’ di Roma) e Center for Life Nanoscience @Sapienza (IIT).
Ricercatori IBPM di riferimento: Pietro Laneve e Elisa Caffarelli
6/12/2019
IBPM a "Più libri più liberi": evento sul DNA - GUARDA IL VIDEO
Documents: PLPL_2019_PROGRAMMA_CNR.pdf
07/11/2019
Nel progetto di alternanza scuola-lavoro svolto nel laboratorio di Maura Cardarelli e Patrizia Brunetti all'IBPM, gli studenti del liceo Pascal imparano tecniche "verdi", basate sullo studio della biologia molecolare delle piante, per decontaminare i terreni dall'arsenico
 Fonte: Rassegna Stampa CNR
Fonte: Rassegna Stampa CNR 08/10/2019
Il premo Nobel 2019 in Fisologia o Medicina per la scoperta dell'adattamento delle cellule alla mancanza di ossigeno
Premi Nobel, i commenti degli esperti Cnr
Rassegna stampa 08/10/2019
I ricercatori Luciano Anselmo (Cnr-Isti), Alberto Ferri (Cnr-Ift), Alessandro Giuffré (Cnr-Ibpm) e Maurizio Peruzzini (Cnr-Dsctm) commentano la recente assegnazione dei premi Nobel per la fisica, per la medicina e la fisiologia e per la chimica, rispettivamente assegnati a James Peebles, Michel Mayor e Didier Queloz, a William G. Kaelin Jr, Sir Peter J. Ratcliffe e Gregg L. Semenza e a John B. Goodenough, M. Stanley Whittingham e Akira Yoshino.
Vedi anche: il servizio al Giornale Rai Radio 1 ’Nobel Fisica 2019’ e il servizio di RaiNews24 ‘Nobel Chimica 2019’
 Fonte: https://www.cnr.it/rassegnastampa/19-10/191008/AJ1YUC.tif
Fonte: https://www.cnr.it/rassegnastampa/19-10/191008/AJ1YUC.tif
23/07/2019
Astrobiologia. Il progetto “VITA NELLO SPAZIO”, cui partecipa l’Unità IBPM coordinate da Giovanna Costanzo, finanziato da ASI, Agenzia Spaziale Italiana. Su PLATINUM https://platinum-online.com/, nuova testata distribuita dal Sole 24 ore

10/07/2019
Meeting with delegation from Ikiam University, Ecuador
Documents: Meeting with delegation from Ikiam University.pdf
Meet in Italy for Life Sciences 2019
28/03/2019

News 26/03/2019
12/03/2019
13/02/2019
Durante la mitosi il materiale genetico deve essere ripartito ugualmente tra le due cellule figlie e tutta la cellula si riorganizza per assicurare una corretta segregazione dei cromosomi, mentre molti processi nucleari, quali la trascrizione e il processamento degli mRNA (“splicing”) sono interrotti. Il lavoro di Pellacani et al., dimostra che due proteine coinvolte nello splicing, Sf3A2 e Prp31, non rimangono inattive durante la mitosi, ma svolgono una funzione essenziale per un corretto legame dei cromosomi ai microtubuli del fuso. Lo studio di Pellacani et al. apre la strada a future ricerche volte a identificare altre “moonlighting proteins” coinvolte non solo nei processi di trascrizione o di splicing ma anche nella regolazione della mitosi. Una precisa definizione dei meccanismi molecolari della mitosi è fondamentale per comprendere i processi di carcinogenesi e sviluppare terapie anti-tumorali.
| Mitosi in cellule S2 di Drosophila di controllo (a) e prive di Prp31 (b) o Sf3A2 (c). Sono state usate cellule che esprimono sia tubulina-RFP, che marca i microtubuli del fuso, che Istone-GFP, che marca i cromosomi. Notare che nelle cellule di controllo la divisione cellulare è completata in circa 1 ora, mentre in assenza di Prp31 o Sf3A2 la cellula permane in mitosi per circa 4 ore. |
Ref: ‘A Newly Identified Flower-Specific Splice Variant of AUXIN RESPONSE FACTOR8 Regulates Stamen Elongation and Endothecium Lignification in Arabidopsis’, Roberta Ghelli, Patrizia Brunetti, Nadia Napoli, Angelo De Paolis, Valentina Cecchetti, Tomohiko Tsuge, Giovanna Serino, Minami Matsui, Giovanni Mele, Gianmarco Rinaldi, Gianna Aurora Palumbo, Fabrizio Barozzi, Paolo Costantino, Maura Cardarelli Mar;30(3):620-637 DOI: 10.1105/tpc.17.00840. (http://www.plantcell.org/content/30/3/620)
NUOVO STUDIO SULLA RIVISTA ONCOGENE FINANZIATO DA AIRC. REGINA ELENA E IBPM-CNR: SCOPERTA NELLA REGOLAZIONE DI FATTORI CRUCIALI PER LA FORMAZIONE DEI TUMORI
Ref: HIPK2 and extrachromosomal histone H2B are separately recruited by Aurora-B for cytokinesis., Laura Monteonofrio, Davide Valente, Manuela Ferrara, Serena Camerini, Roberta Miscione, Marco Crescenzi, Cinzia Rinaldo, Silvia Soddu, 2018 Mar 22. https://doi.org/10.1038/s41388-018-0191-6
Fonte: www.cnr.it
Fonte: www.cnr.it
DISTROFIA DI DUCHENNE: IDEATO UN NUOVO GENE ARTIFICIALE A SCOPO TERAPEUTICO